1. INTRODUCCIÓN
En los últimos años hemos podido observar un enorme uso en las Humanidades Digitales de herramientas computacionales y de técnicas estadísticas que han abierto múltiples líneas de investigación y docencia. En el espacio de la Lingüística Histórica la aplicación está siendo más restringida, al menos en el ámbito hispánico2. Sin embargo, esta metodología viene siendo usada sistemáticamente y avalada desde hace al menos una década por múltiples especialistas (Holman et al., 2008; Longobardi & Guardiano, 2009; Sicoli & Holton, 2014; Jäger & List, 2015; Jäger & Wichmann, 2016). En algunos casos, incluso, se ha utilizado la estadística para intentar reconstruir lenguas antiguas (Bouchard-Côté et al., 2013).
La base del procedimiento es el paralelismo que existe entre los procesos del cambio lingüístico y la evolución de las especies. El lingüista y el biólogo evolutivo, a menudo, se encuentran ante problemas y preguntas similares (Boc et al., 2010, p. 647). El uso de herramientas computacionales no excluye en ningún caso la labor del especialista, que ha de filtrar, analizar y justificar los resultados que nos puede ofrecer una estimación filogenética. El método comparativo tradicional tiene múltiples beneficios, pero su aplicación es lenta y requiere a lingüistas muy formados (Jäger & List, 2016), por ello, no es extraño el giro hacia los análisis cuantitativos y el uso de la lingüística computacional.
El objeto de este artículo es explicar y analizar el uso de una de las herramientas más conocidas y con mayor presencia en el ámbito de la filogenia y promover el uso de la misma, así como incentivar el uso de la ciencia estadística en el marco de la enseñanza filológica3.
2. QUÉ ES PAUP*
PAUP*4 (Swofford, 2002) es un programa de análisis filogenético desarrollado en 1993 por David Swofford y probablemente uno de los programas de análisis más utilizado en la actualidad. Es un software comercial que requiere una licencia de uso de pago. Inicialmente permitía realizar análisis filogenéticos usando el procedimiento de parsimonia, de ahí sus siglas Phylogenetic Analysis Using Parsimony. Posteriormente, Swofford incorporó otros métodos de análisis estadístico como máxima verosimilitud o el análisis de distancias, añadiendo al nombre el asterisco con el significado and other methods.
El programa posee una gran variedad de opciones de análisis y selección de modelos, y permite evaluar distintos tipos de información desde cadenas de ADN, ARN, y, como tendremos ocasión de comprobar, otro tipo de datos. Además, tiene distintas opciones para trabajar con árboles filogenéticos entre las que se incluyen su importación, combinación, comparación, enraizado, evaluación de hipótesis, etc.
PAUP* está disponible en versiones para distintas plataformas como Macintosh5, Windows o Linux. Por el momento está disponible una versión gratuita sin límite temporal de uso, probablemente hasta el próximo desarrollo, según indica en la página del proyecto.
3. LOS ARCHIVOS NEXUS
PAUP* trabaja con archivos Nexus. Se trata de un sistema de datos usado en bioinformática, consistente en ficheros de texto organizados y etiquetados de un modo concreto. Estos pueden almacenar múltiples datos sobre taxones, distancias, etc. e incluso árboles de clasificación. El formato de archivo no es exclusivo de PAUP* y podemos trabajar con estos archivos también en otros programas de análisis como MrBayes o Mesquite.
Ofrecemos a continuación un ejemplo de archivo Nexus, entre corchetes indicamos la función de cada uno de los elementos:
#NEXUS
BEGIN TAXA;
DIMENSIONS NTAX=5 [define el tamaño del alineamiento, en este caso 5 taxones]
TAX LABELS Especie1 Especie2 Especie3 Especie4 Especie5; [Etiquetas de los taxones]
END;
BEGIN CHARACTERS;
DIMENSIONS NCHAR=15 [número de caracteres en cada uno de los taxones]
FORMAT DATATYPE=DNA missing=? gap=-; [Define el tipo de datos (DNA) y los símbolos para los datos ausentes y los espacios (?)y (-)]
MATRIX [Aquí comienza el alineamiento de datos...]
Especie1 atgctagctagctcgagcta
Especie2 atgcta??tag-tagagctb
Especie3 atgttagctag-tggagcta
Especie4 atgttagctag-tag--?aa
Especie5 atgvtaa-??—gatagctab;
END; [Final del bloque de datos]
El formato Nexus fue desarrollado por David Maddison, Wayne Maddison y el propio David Swofford para facilitar el intercambio de archivos entre los programas usados en filogenia, creando un único tipo de archivo (Maddison et al., 1997, p. 590).
Una de las grandes ventajas de los archivos Nexus es su modularidad y la capacidad de incorporar múltiples tipos de datos. Pero sin duda la característica que ha convertido este tipo de archivos en un estándar es que pueden ser analizados y editados desde cualquier editor o procesador de texto.
4. ARCHIVOS NEXUS CON DATOS LINGÜÍSTICOS
Al igual que podemos establecer árboles evolutivos de organismos con métodos algorítmicos y estadísticos, podemos extrapolar dicha metodología al análisis de lenguas, la clave es cómo crear las cadenas de datos para que sean analizadas (Wichmann & Saunders, 2007, pp. 375-379).
Para entender cómo podemos desarrollar archivos Nexus con datos lingüísticos tenemos que hablar del trabajo de M. Swadesh y de la lista que desarrolló para intentar delimitar el cambio lingüístico. Realizó una lista de vocabulario básico resistente a préstamos, formado por palabras comunes existentes en cualquier lengua. Inicialmente su listado incluía 200 términos, que fueron reducidos a 100 posteriormente (Swadesh, 1955).
Según Swadesh, el listado permite mediante la comparación establecer la relación entre dos lenguas. Su propuesta parte de la premisa de que en el léxico de cualquier lengua podemos encontrar un vocabulario estable y que se ha mantenido sin cambios. A partir de este punto, es necesario asumir que existe una tasa de retención del léxico de manera constante. De este modo, determinado vocabulario que podríamos considerar como básico (partes del cuerpo, acciones primarias, familia, etc.) persiste en el tiempo (Swadesh, 1960, p. 133).
Partiendo del porcentaje de cognados compartidos entre dos lenguas Swadesh sostiene que podemos computar el tiempo transcurrido desde que se separaron. De este modo, plantea una constante glotocronológica, la cual establece un ritmo de cambio fijado en un promedio del 14% cada 1000 años (constante de 86%).
Este intento de aplicar principios matemáticos a listados lingüísticos es enormemente problemático ya que podemos encontrarnos con préstamos entre lenguas y no con cognados reales con un mismo origen, o con que los cognados sean irreconocibles debido al paso del tiempo y a las mutaciones lingüísticas que han ido ocurriendo. O incluso podría ser el caso que dicho vocabulario universal pudiese no existir (Coseriu, 1965, p. 92).
Sin embargo, la propuesta de Swadesh será recogida por el grupo de trabajo de Michael Dunn en 2012 en su base de datos Indo-European lexical cognacy database (IELEX)6, actualmente alojada en Dunn & Tresholdi (2021).
La principal diferencia es que amplían significativamente el número de lenguas objeto de análisis, elevándolo a 163, y trabajando con un conjunto de 34619 palabras y con un total de 32651 caracteres codificados7. y se abrirá trabajo tuvo una exposición mediática notoria, ya que, con el análisis de los datos del cambio lingüístico, decantaban la balanza por la hipótesis anatolia de C. Renfrew sobre el origen de las lenguas indoeuropeas. (Bouckaert et al., 2012). Asumiendo que el cambio lingüístico es constante y, de este modo, datar el origen de las lenguas indoeuropeas con la tesis antes mencionada (Renfrew, 1990).
Posteriores estudios (Chang et al. , 2015), sin embargo, retornaban a la hipótesis de las estepas con un análisis filogenético similar que incluía restricciones en las familias de lenguas.
Los repositorios de datos en el sitio del proyecto original no están disponibles, pero el propio M. Dunn y T. Tresoldi han archivado parte de la información del proyecto IELEX (Dunn & Tresoldi, 2021). Facilitando el acceso a los archivos Nexus que el proyecto desarrolló y posibilitando la descarga del archivo ielex.nex en dicho vínculo.
Para entender la composición del archivo del proyecto es necesario explicar la novedad que incorpora el proyecto IELEX. La clasificación de conceptos se realiza en función de las clases de cognados que se van delimitando para cada palabra. De este modo, el concepto padre se relaciona en las diversas lenguas indoeuropeas con distintas clases de cognados, como podemos ver en la siguiente tabla 1, en la que se delimitan ocho posibles cognados en torno a los cuales se clasifican las lenguas:
|
Clase A |
Armenio, hayr; asamés, pitā; baluchi, phith; catalán, pare; danés, fader; neerlandés, Vader; inglés, father; francés, père; friulano, pari; alemán, Vater; griego, pateras; guyaratí, pita; hindi, pita; islandés, faðir; irlandés, athair; italiano, padre; cachemir, bab; maithili, pitā; noruego, far; pastún, plar; persa, pedar; portugués, pai; gaélico-escocés, athair; español, padre; sueco, fader; tayiko, padar; zazaki, pī. |
|
Clase B |
Checo, otec; macedonio, otec; polaco, ojciec, ruso, отец, servo-croata, otac, eslovaco, otec, esloveno, oce. |
|
Clase C |
Albanés baba; bengalí baba, marathi bap, nepalí babu, oriya, bapa, shindi, bābō, urdu, باپ |
|
Clase D |
Búlgaro tatko, letón tę̃vs, lituano tevas, rumano, tată, galés, tad |
|
Clase E |
Bieloruso bac'ka, ucraniano, bat'ko |
|
Clase F |
Luxemburgués, papp |
|
Clase G |
Lahnda. Pyu, panjabí, pyo, sinhala, piya |
|
Clase H |
Sardo, babbu |
El paso siguiente es interpretar los datos como binarios. Para ello cada clase de cognado se convierte en un carácter con dos valores válidos, 0 y 1. Siendo 0 el valor que se da a una lengua que no usa un concepto de la clase de cognado y 1 el valor que recibe al usar esa clase de cognado. El archivo que crean incorpora 94 taxones y cadenas de 3937 caracteres correspondientes a los elementos coincidentes y no coincidentes.
De este modo, ya tenemos para cada lengua objeto de análisis un conjunto de caracteres que proporcionan los datos que PAUP* puede interpretar.
5. ANÁLISIS CON PAUP*
Una vez abierto el archivo que va a ser analizado, PAUP* nos indica el tamaño del mismo, el número de elementos y los caracteres presentes en cada uno de ellos (la información que aparece en la cabecera de nuestro archivo Nexus). Podemos comenzar con una búsqueda heurística (>Hsearch), que tratará de hallar coincidencias siguiendo el criterio que tenemos por defecto, en este caso parsimonia, pero que podrá ser modificado en cualquier momento. De este modo y una vez concluida esta acción heurística, PAUP* nos indica el número de árboles obtenidos y el tiempo de cálculo utilizado. En este caso, podemos grabar todos los árboles (>SaveTrees file='NOMBREARCHIVO.tree' format=Newick). O bien, podemos indicar a PAUP* que realice una síntesis del árbol más probable a partir de los obtenidos (>Contree). Si no especificamos un tipo de consenso, el árbol por defecto será de estricto consenso, pero también podemos escoger un criterio de consenso de mayoría8.
Hemos indicado el nombre del archivo y el formato en el que lo hemos grabado es Newick. El formato de árbol Newick es una manera de representar utilizando paréntesis y comas. Fue adoptado por un grupo de investigadores9 entre los que se encontraba el propio D. Swofford, autor de PAUP*. Además posee algunas capacidades gráficas y es posible presentar en pantalla todos los árboles obtenidos en la búsqueda (>showtrees all), o, individualmente, utilizando un número correspondiente a los árboles obtenidos en el anterior análisis. El directorio de trabajo en el que se grabará los datos será el mismo desde el que hemos cargado nuestro archivo Nexus.
Anteriormente hemos mencionado que podemos cambiar el método de análisis de máxima parsimonia (MP) por otro tipo de criterio, como el de máxima verosimilitud (Maximum Likelihood o ML) o el de distancia (Distance). Cada uno de ellos posee unas características propias y pueden ser útiles dependiendo de los análisis que estemos realizando10. El cambio al criterio de ML puede realizarse a través de la línea de comando (>set criterion=likelihood) o bien a partir del sistema de menús implementado. Y, a continuación, podemos volver a lanzar una búsqueda heurística de coincidencias con el comando Hsearch ya conocido. Dependiendo de la rapidez de nuestro ordenador el proceso podrá durar más o menos11 y se abrirá una pestaña que nos indicará el estado de este. De nuevo podemos introducir el comando >showtrees para ver el resultado obtenido con este tipo de análisis estadístico.
Las primeras versiones de PAUP* permitían solamente la introducción de datos mediante la línea de comandos, como hemos ido mostrando en nuestros ejemplos, sin embargo, en las últimas versiones, D. Swofford ha implementado la opción de ventanas y menús que permiten la navegación y selección de las opciones sin necesidad de recurrir a la entrada por teclado. De este modo, podemos cambiar el análisis en el menú superior (Analysis) y elegir, de este modo, el tipo que queremos realizar. También podemos cambiar las opciones para cada uno de ellos, posibilitando diferentes metodologías. Igualmente, en el menú Trees podremos acceder a los distintos árboles que se han generado con nuestro análisis. Desde aquí también podremos acceder a nuestros árboles e imprimirlos o convertirlos en un archivo PDF.
Como forma de representación PAUP* permite escoger bien un cladograma o un filograma; el primero no cambia cuando se escoge una forma de reconstrucción diferente, sin embargo, el filograma sí lo hace. El filograma dibuja las longitudes de las ramas en proporción al número de cambios inferidos en la reconstrucción (>describetree /plot=phylo).
6. REVISIÓN DE LOS DATOS
Los árboles generados con uno y otro método nos proporcionan un material susceptible de ser analizado a través de la lingüística clásica, determinando el acierto o error de este tipo de análisis. A continuación, reproducimos el árbol de lenguas indoeuropeas generado con PAUP* a partir de un análisis de MP. Para computar el soporte de cada clado se realiza un bootstrap (addseq=random, nreps=100)12. El análisis generó 69 árboles. El árbol de consenso con una longitud de 429413, un Índice de consistencia14 (CI) de 0,3912 y un índice de retención15 (RI) de 0,6184.
Un procedimiento que proporciona PAUP* con el que podremos ver la consistencia del árbol resultante es el ya mencionado bootstrap. Este proceso básicamente realiza mutaciones aleatorias de cada elemento (cada lengua en nuestro caso) y reconstruye el árbol sobre los datos mutados para ver la consistencia de las ramificaciones en el árbol inicial. El proceso en PAUP podemos repetirlo n veces y PAUP* reporta un árbol donde a cada rama se le asigna un número entre 0 y 100, que sería el tanto por ciento de iteraciones donde esa rama se ha producido. Las más fiables son las que se mantienen a partir del 90%, ya que las mutaciones aleatorias no llegan a alterar las diferencias con otros elementos hasta el punto de clasificarlo en una rama diferente.
El bootstrap es un método usado con frecuencia en MP y ML y en PAUP* podemos indicar el número de repeticiones que deseamos (lo más común es otorgar un valor de 10016).

Los grupos de familias se muestran perfectamente delimitados. Por ejemplo, el soporte 100 de bootstrap en la rama céltica, la rama germánica, la latina o la eslava así nos lo atestiguan De hecho, se observa un súper clado con un elevado soporte entre las ramas armenio-griega, germánica, latina, céltica y eslava. Sin embargo, el análisis muestra algún problema en la interpretación de las lenguas germánicas antiguas, tal y como vemos al introducir en el mismo nodo el gótico, el alto alemán antiguo y el antiguo inglés o anglosajón.
La premisa de los análisis de parsimonia es que las mutaciones son ocasionales y raras, y en el caso de la evolución lingüística, el resultado puede estar falseado cuando se han producido múltiples cambios. De hecho, ramas caracterizadas por múltiples cambios tienden a estar más próximas de lo necesario en los análisis de parsimonia. Se trata del fenómeno al que Felsenstein denomina “long branch attraction” (2004, p. 122). De este modo, el análisis de parsimonia será más certero con lenguas no excesivamente divergentes y con una evolución reducida (Wichmann & Saunders, 2007, p. 388). Probablemente, el análisis de ML nos ofrezca mejores resultados, pero el tiempo de computación es sensiblemente más elevado. Una vez modificado el criterio de análisis a likelihood, podemos optimizar algunos parámetros tal y como hace G. Jäger (2016)17. El resultado que obtenemos es el siguiente:
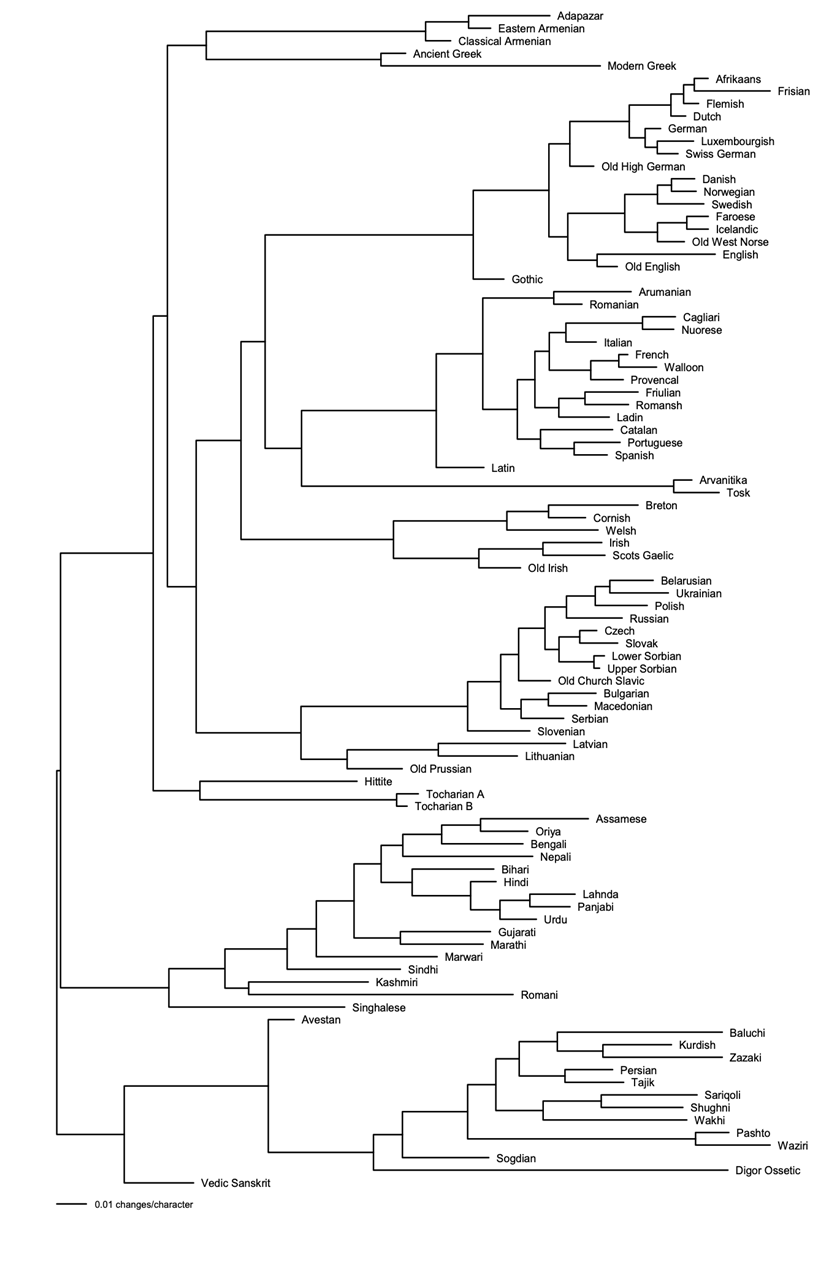
En este caso el árbol generado interpreta bastante mejor las lenguas antiguas, introduciéndolas en el mismo nodo que sus lenguas descendientes, pero marcando los cambios con la separación de los nodos secundarios. Los grandes grupos lingüísticos están perfectamente diferenciados y delimitados y las lenguas antiguas están perfectamente asociadas. Volvemos a encontrar indicios del súper clado que aglutinaría lenguas eslavas, célticas, latinas y germánicas.
Sin embargo, podemos constatar algunos contrastes evidentes. El alto alemán antiguo aparece también como lengua emparentada con el grupo bajo alemán constituido por el afrikáans, el flamenco, el frisón o el neerlandés. Un error más que evidente que podría deberse a la falta de un taxón específico de bajo alemán antiguo, que habría podido desligarlo.
Un patrón que debemos considerar también es la consideración de los estadios antiguos de algunas lenguas y su relación con sus descendientes. El alto alemán antiguo aparece no como antecedente del alemán, sino que está situado en una rama paralela; en términos familiares, la representación de estas lenguas sería una relación nepotal18, cuando la relación sabemos que es directa.
Este patrón lo podemos ver repetido con el nórdico antiguo19 o el inglés antiguo20 e incluso con lenguas no indoeuropeas, y posiblemente se deba a innovaciones producidas en las lenguas descendientes que no se han dado en el ancestro común (Garrett, 2018, p. 33). La solución pasa por implementar restricciones que limiten los posibles árboles a aquellos en los que el alto alemán antiguo aparezca como antecedente del suizo alemán, del alemán moderno o el luxemburgués.
Dichas restricciones podemos realizarlas con el comando constraints, con él podemos definir los grupos que tenemos la certeza de que están asociados21. Podemos forzar la búsqueda de árboles compatibles con esta restricción y de este modo filtrar los resultados a únicamente los que incorporan la misma (>hsearch constraints=Old_High_German enforce=yes)
Es evidente que la labor del lingüista es indispensable no solamente en el análisis de los resultados, sino también en el proceso de obtención de estos. En ese sentido la labor de revisión del especialista se hace indispensable para la obtención de resultados (Chang et al., 2015, p. 199). Este tipo de restricciones serán las que incorporen Chang et al. (2015) en su estudio, restringiendo ocho lenguas antiguas y medievales y convirtiéndolas en el ancestro de 39 lenguas descendientes (Chang et al., 2015, p. 199). El resultado es un nuevo soporte para el origen del indoeuropeo en las estepas, en el que se modifica radicalmente la datación de los estudios de Bouckaert et al. (2012).
Un aspecto que no debemos dejar de lado es la flexibilidad que nos permite el tipo de archivo Nexus. De este modo, podemos editar el archivo completo que hemos venido utilizando con cualquier editor de textos y limitarnos al grupo de lenguas que nos interese, teniendo en cuenta que es necesario redefinir el número de etiquetas y taxones en la cabecera del archivo. El reajuste reducirá sensiblemente los tiempos de computación y facilitará en gran manera el uso de otras herramientas que nos proporciona PAUP*22.
En este caso concreto hemos creado un archivo Nexus con 22 taxones correspondientes a las lenguas germánicas y al que se ha añadido también el protoindoeuropeo23 y los tiempos de computación se reducen ostensiblemente. Al realizar un análisis de ML y su consiguiente análisis de bootstrap podemos observar lo siguiente:
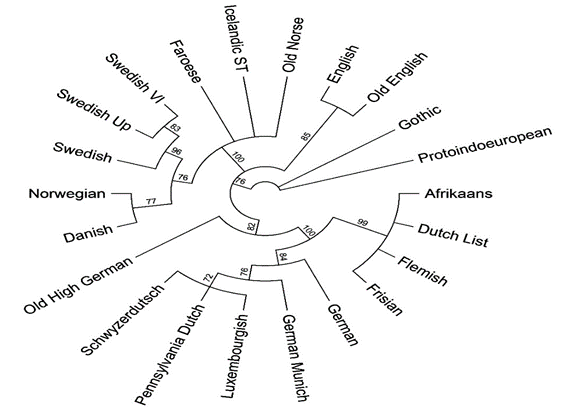
Para el ejemplo expuesto, hemos generado un análisis de bootstrap con 100 réplicas y 10 secuencias aleatorias (bootstrap nreps=100/addseq=random nreps=10). El soporte para diferenciar las lenguas separadas por la segunda mutación consonántica es el máximo. Pero no hay un soporte para el alto alemán antiguo (por defecto un soporte inferior a 70 no aparece en el gráfico). Hemos enraizado el árbol a partir del protoindoeuropeo y observamos la delimitación de las lenguas germánicas orientales y un grupo que aglutinaría las occidentales y nórdicas. La posición del nórdico antiguo sería incierta a partir de los datos del bootstrap. El soporte para un grupo formado por las lenguas alto y bajo alemanas también sería el más alto; del mismo modo que el que aparece para las lenguas nórdicas (en ambos casos un bootstrap 100).
PAUP* nos permite, como vemos, una enorme flexibilidad, no solamente a la hora de establecer los criterios de análisis, como ocurre al fijar el margen de soporte de nuestro estudio; sino también al editar los archivos NEXUS donde nos permite incluir grupos de lenguas, como por ejemplo las latinas o las célticas, con el fin de delimitar mejor las distancias o los grupos a partir de la raíz original. En ese sentido, tal y como se realiza con frecuencia en análisis de ADN o ARN, la introducción de cadenas bien diferenciadas puede clarificar los árboles resultantes. De este modo, hemos elaborado un archivo NEX con 40 taxones que incluyen lenguas germánicas, latinas y célticas y, con él, procedemos a realizar un análisis de ML.
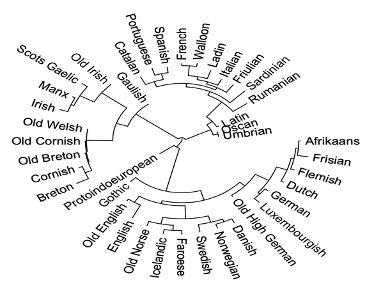
Lo siguiente es evaluar la consistencia del árbol obtenido mediante el proceso de bootstrap, para el que hemos escogido unos valores de 100 repeticiones y un límite de 80. Por tanto, no se indicarán las consistencias inferiores a ese valor.
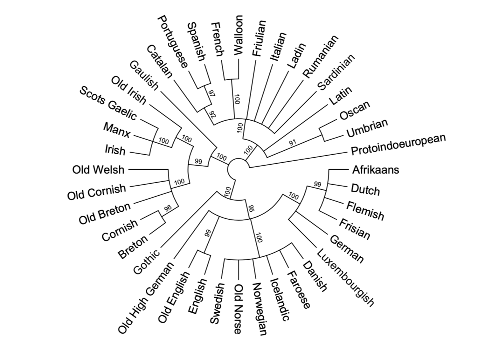
Observamos que el soporte de los grupos lingüísticos ha subido al máximo; en ese sentido, si lo comparamos con el anterior bootstrap (Figura 4), al introducir los contrastes con los grupos céltico y latino, el respaldo al análisis se ha incrementado en la distinción del grupo del germánico occidental y del grupo nórdico. También es necesario señalar que se ha aplicado otro refinamiento al árbol resultante, ya que hemos enraizado todo el árbol a partir del grupo indoeuropeo (>roottrees). Las posiciones de los estadios lingüísticos más antiguos, como el latín, el galo, el gótico o el alto alemán antiguo están adscritos correctamente a sus grupos correspondientes, sin embargo, su posición es incierta.
Anteriormente mencionábamos que en algunos casos un análisis de MP podría ser conveniente cuando las mutaciones entre los taxones no eran frecuentes. En ese sentido, hemos hallado algunos indicios en los análisis realizados de un súper clado que aglutinaría los grupos lingüísticos latino, céltico y germánico y, de este modo, podría ser una buena opción para este caso concreto.
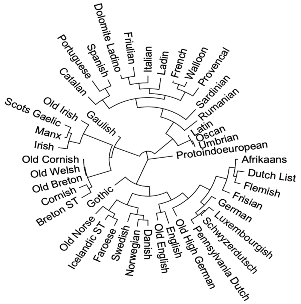
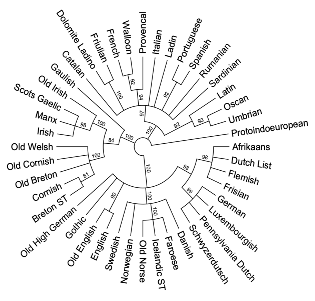
De nuevo observamos los grupos claramente identificados, pero en el caso concreto de las lenguas germánicas se delimitan perfectamente los grupos oriental, occidental y septentrional.
7. CONCLUSIÓN
El uso de herramientas de cladística proporciona indudables ventajas en la representación y en el análisis de árboles lingüísticos, tal y como hemos tenido ocasión de demostrar. Sin embargo, coincidimos con Rexová et al. (2003, p. 120) en que la barrera entre humanidades y ciencias es probablemente el principal motivo por el que la metodología de análisis cladístico no ha sido introducida definitivamente en la lingüística comparada.
El proyecto IELEX y los archivos que desarrollaron han marcado la línea a seguir en el campo de la Lingüística Histórica Computacional, y su testigo ha sido recogido por el proyecto IE-COR, Indo-European Cognate Relationships dirigido por el Max Planck Institute for Evolutionary Anthropology. Su trabajo incrementa y mejora las bases de datos originales de IELEX, pero todavía no hay ningún dato publicado, y el uso de las bases de datos está restringido a los investigadores del proyecto. En ese sentido, la principal limitación con la que contamos es la escasez de archivos que permitan el análisis cladístico. En estos momentos contamos con los desarrollados por Dyen et al. (1997) y que fueron retomados por el proyecto IELEX. De hecho, los archivos Nexus desarrollados por IELEX son básicamente la única fuente con la que podemos contar. Al menos hasta que el proyecto IE-COR haga públicas sus bases de datos.
La línea que siguió el proyecto IELEX en su base de datos fue la de establecer la relación lingüística de las lenguas a partir del análisis de los cognados. Esto provoca una serie problemas en los árboles resultantes. La relación que se establece entre el alto alemán antiguo y las lenguas bajo alemanas, por mencionar uno de los ejemplos más claros. Las relaciones semánticas no toman en cuenta las mutaciones consonánticas, que serían un criterio también clave en la delimitación. Estas carencias serán las que intente suplir el proyecto ASJP que usará un número más reducido de conceptos, y se centrará en crear una base de datos a partir de 41 posibles sonidos. De este modo, y basándose en la distancia Levenshtein crearán una serie de matrices de distancias entre lenguas.
Hemos destacado ya anteriormente las posibilidades que abre esta herramienta en la docencia, en nuestro caso concreto de Historia de la Lengua Alemana. El uso de este tipo de recursos en el aula permite la incorporación de elementos de análisis estadístico y lingüística computacional a la disciplina tradicional y proporciona a los estudiantes una forma de acceso clara y sencilla a las clasificaciones de lenguas y al análisis de estas.
Otra de las cuestiones que tenemos que plantearnos es qué grado de confianza podemos dar a los datos que nos proporcionan los programas de análisis filogenético como PAUP*. La clave para obtener una respuesta nos la ha de dar la comparación con los métodos tradicionales (Wichmann, & Saunders, 2007, p. 400) y por tanto la labor del lingüista es indispensable a lo largo de todo el proceso, constituyendo el proceso de la lingüística comparada y el estadístico, desarrollos complementarios. En palabras de G. Jäger y List:
To summarize, the intellectual goals of the comparative method and of modern computational historical linguistics overlap, but they are not identical. To formulate it in a pointed way, the comparative methods strive to reconstruct the true history of languages in their entirety while statistical approaches search for probable or at least useful models of the observed patterns in some well-defined partial range of data. Despite these differences, they can benefit from each other. Computational approaches utilize the findings of the comparative method both as raw data and as gold standard to validate their findings. Conversely, computational approaches are well-suited to generate initial hypotheses especially about understudied languages, to be evaluated manually by human experts (2016, p. 30).
REFERENCIAS BIBLIOGRÁFICAS
Ben Hamed, M., Darlu, P., & Vallée, N. (2003). On Cladistic Reconstruction of Linguistics Trees through Vowel Data. Journal of Quantitative Linguistics, 12, 79-109. https://doi.org/10.1080/09296170500055467
Boc, A., Sciullo, A. M. di, & Makarenkov, M. (2010). Classification of The Indo-European Languages Using a Phylogenetic Network Approach. En H. Locareck-Junge, & C. Weihs (Eds.), Classification as a Tool for Research (pp. 647-655). Springer. https://doi.org/10.1007/978-3-642-10745-0_71
Bouchard-Côté, A., Hall, D., Griffiths, T. L., & Klein, D. (2013). Automated Reconstruction of Ancient Languages Using Probabilistic Models of Sound Change. Proceedings of the National Academy of Sciences, 36(2), 141-150. https://doi.org/10.1073/pnas.1204678110
Bouckaert, R., Lemey, P., Dunn, M., Greenhill, S. J., Alekseyenko, A. V., Drummond, A. J., Gray, R. D., Suchard, M. A., & Atkinson, Q. D. (2012). Mapping The Origins and Expansion Of The Indo-European Language Family. Science, 337, 957-960. https://doi.org/10.1126/science.1219669
Chang, W., Cathcart, Ch., Hall, D., & Garrett, A. (2015). Ancestry-Constrained Phylogenetic Analysis supports the Indo-European Steppe Hypothesis. Language (Baltim), 91, 194-244. https://doi.org/10.1353/lan.2015.0005
Coseriu, E. (1965). Critique de la glottochronologie appliquée aux langues romanes. En G. Straka (Ed.), Linguistique et Philologie Romanes. Xe Congrès International de Linguistique et Philologie Romanes. Klincksieck, 87-96.
Dunn, M., & Tresoldi., T. (2021). evotext/ielex-data-and-tree: IELex data and tree (Version r20211108) [Data set]. Zenodo. https://doi.org/10.5281/zenodo.5556801
Dyen, I., Kruskal, J. B., & Black, P. (1997). Comparative IE Database Collected by Isidore Dyen. http://www.ntu.edu.au/education/langs/ielex/IE-RATE1
Felsenstein, J. (2004). Inferring Phylogenies. Sinauer Associates.
Garrett, A. (2018). New Perspectives on Indo-European Phylogeny and Chronology. Proceedings of the American Philosophical Society, 162(1), 25-38.
Jäger, G. (2016). Phylogenetic trees IV. Maximum Likelihood [presentación]. Computational Historical Linguistics, ESSLLI 2016 (European Summer School in Logic, Language and Information), Bolzano-Bozen. https://www.sfs.uni-tuebingen.de/~gjaeger/lehre/esslli2016/slides/maximumLikelihood.pdf
Jäger, G., & List, J. M., (2015). Factoring Lexical and Phonetic Phylogenetic Characters from Word Lists. En H. Baayen, G. Jäger, M. Köllner, J. Wahle, & A. Baayen-Oudshoorn (Eds.), Proceedings of the 6th Conference on Quantitative Investigations in Theoretical Lingusitics. https://doi.org/10.15496/publikation-8625
Jäger, G., & List, J. M. (2016), Statistical and Computational Elaborations of The Classical Comparative Method. http://www.sfs.uni-tuebingen.de/~gjaeger/publications/jaegerListOxfordHandbook.pdf
Jäger, G., & Wichmann, S. (2016). Inferring the World Tree of Languages from Word Lists. En S. G. Roberts, C. Cuskley, L. McCrohon, L. Barceló-Coblijn, O. Fehér, & T. Verhoef (Eds.), The Evolution of Language: Proceedings of the 11th International Conference (EVOLANGX11). http://evolang.org/neworleans/papers/147.html
Holman, E. W., Wichmann, S., Brown, C. H., Velupillai, V., Müller, A., & Bakker, D. (2008). Advances in Automated Language Classification. En A. Arpe, K. Sinnemäki, & U. Nikkan (Eds.), Quantitative Investigations in Theoretical Linguistics (pp. 40-43). University of Helsinki.
Longobardi, G., & Guardiano, C. (2009). Evidence for Syntax as A Signal Of Historical Relatedness. Lingua, 119(11), 1679-1706. https://doi.org/10.1016/j.lingua.2008.09.012
Maddison, D., Swofford, D., & Maddison, W. (1997). Nexus: An Extensible File Format For Systematic Information. Systematic Biology, 46, 590-621.
Mayr, E. (1974). Cladistic analysis or cladistics classification? Journal of Zoological Systematics and Evolutionary Research, 12, 94-128. https://doi.org/10.1111/j.1439-0469.1974.tb00160.x
Muscio, H. J. (2010). Transferencia Horizontal, Cladismo y Filogenias Culturales, Clasificación y Arqueología. En D. García Rivero, & J. L. Escacena Carrasco (Eds.), Enfoques y Métodos Taxonómicos a la Luz de la Evolución Darwiniana (pp. 223-251). Prensa de la Universidad de Sevilla.
Peña, C. (2011). Métodos de inferencia filogenética. Revista Peruana De Biología, 18(2), 265-267. https://10.15381/rpb.v18i2.243
Peñarrubia Navarro, P. (2021). Estilometría con fines geolingüísticos aplicada al corpus COSER, Revista de Humanidades Digitales, 6, 22-42. https://doi.org/10.5944/rhd.vol.6.2021.30870
Renfrew, C. (1990). Archaeology and Language. The Puzzle of the Indo-European Origins. Cambridge University Press.
Rexová, K., Frynta, D., & Zrzavý, J. (2003). Cladistic Analysis of Languages: Indo-European Classification Based on Lexicostatistical Data. Cladistics, 19, 120-127.
Ringe, D., Warnow, T. Taylor A., & Clackson, J. (2002), Indo-European and Computational Cladistics. Transactions of the Philologicval Society, 100:1, 59-129.
Ringe, D. (2012), Cladistic principles and linguistic reality: the case of West Germanic. En Porbert Ph. y Willi A., Laws and Rules in Indo-European, Oxford University Press, 33-42. https://10.1093/acprof:oso/9780199609925.003.0003
Semple, C., & Steel, M. (2003). Phylogenetics. Oxford University Press.
Sicoli, M. A., & Holton, G. (2014). Linguistic Phylogenies Support Back-Migration from Beringia To Asia. PloS One, 9(3), e91722.
Svennblad, B., Erixon, P., Oxelman, B., & Britton, T. (2006). Fundamental Differences Between the Methods of Maximum Likelihood and Maximum Posterior Probability in Phylogenetics. Systematic Biology, 55(1), 116-121.
Swadesh, M. (1955). Towards Greater Accuracy in Lexicostatistic Dating. International Journal of American Linguistics, 21, 121-137.
Swadesh, M. (1960). Tras la huella lingüística de la prehistoria. UNAM.
Swofford, D. (2002). PAUP*. Phylogenetic Analysis Using Parsimony (* and other methods). Sinauer Associates. [Computer software]. https://paup.phylosolutions.com/
Wichmann, S., & Saunders, A. (2007). How to Use Typological Databases in Historical Linguistic Research. Diachronica, 24(2), 373-404. https://doi.org/10.1075/dia.24.2.06wic
Wichmann, S., Holman, E. W., & Brown, C. H. (Eds.). (2020). The ASJP Database (version 19). https://asjp.clld.org
paup> lset basefreq = estimate [para ver las frecuencias base de cada taxón]
paup> lset rates=gamma shape = estimate [rangos de distribución gamma]
paup> lset pinvar=estimate [PInvar para especificar la proporción de sitios invariables, que no aceptan sustituciones]
paup> hsearch [inicia la búsqueda heurística]
paup> describetree /plot=phylo [muestra el árbol con una longitud de ramas en proporción directa al número de cambios asignado a cada rama]
lscores /aic=yes [calcula la similitud de los árboles y el AIC].

 https://orcid.org/0000-0001-6527-203X
https://orcid.org/0000-0001-6527-203X